PROG0301 - Single-nucleotide polymorphism
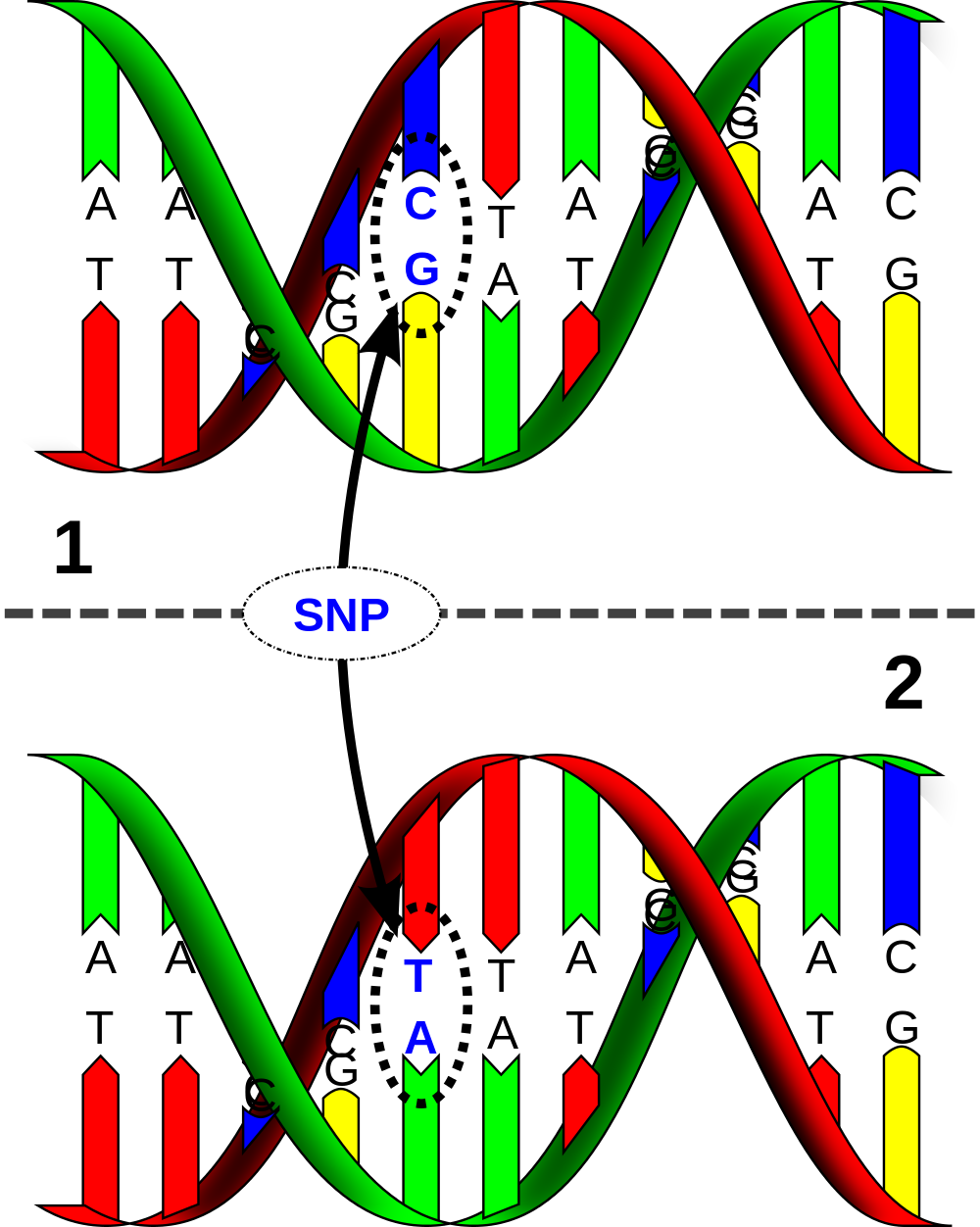
A single-nucleotide polymorphism (abbreviation: SNP; pronunciation: snip) is a DNA sequence variation — a polymorphism — occuring when a single nucleotide (A, C, G or T) in the genome (or another shared sequence) differs between two members of a biological species or paired chromosomes in a human. For example, the two sequenced DNA fragments from different individuals, AAGCCTA to AAGCTTA, contain a difference in a single nucleotide at the 5th position. In this case we say that there are two alleles.
These genetic variations underlie differences in our susceptibility to certain diseases. The severity of illness and the way our body responds to treatments are also manifestations of genetic variations. For example, a single mutation in the APOE (apolipoprotein E) gene is associated with a higher risk for Alzheimer disease.
Assignment
In this exercise, DNA sequences are represented as strings that only contain the upper case letters A, C, G and T, representing the individual nucleotides.
- Write a function SNP that takes two DNA sequences as its arguments. In case these sequences have the same length and only differ at a single location, the function must return a tuple containing three elements. The first element gives the position of the nucleotide that differs between both sequences (positions are indexed from 0). The second and third element are the nucleotides occuring at that position in respectively the first and the second DNA sequence. Otherwise, the function should return the value None.
- Searching for SNPs is usually done by scanning the entire genome (or a long DNA fragment) of an individual to compare it with a read (a shorter DNA fragment) of another individual. A SNP is found at a particular position in the genome if all corresponding nucleotides of the read are the same except for a single one. The position of the diverging nucleotide is then used as the position of the SNP. Of course it is possible that multiple SNPs are found when scanning the genome against a single read. An illustration of this is shown in the following picture. Use the function SNP to write a function SNPs, that takes two arguments: the DNA sequence of a genome and the DNA sequence of a read. The function must return a list containing the positions of all SNPs that are found when comparing the genome against the read. Indexing of the genome positions starts at 0 and the returned list should contain the SNP positions in increasing order.
Example
>>> SNP('AAGCCTA', 'AAGCTTA') (4, 'C', 'T') >>> SNP('AAGCCTAA', 'AAGCTTA') >>> SNP('AAGCTTA', 'AAGCTTA') >>> SNP('AAGCCCA', 'AAGCTTA') >>> SNPs('AGCTGATAAGCCTAAGCGCT', 'AAGCTTA') [11] >>> SNPs('ATCGTAAGCCTAAGGCTACGCTTAGAGATA', 'AAGCTTA') [9, 18] >>> SNPs('AAGCCTAAGCCTA', 'AAGCTTA') [4, 10]
Een enkel-nucleotide polymorfisme (engels: single nucleotide polymorphism; afgekorting: SNP; uitspraak: snip) is een variatie in het DNA — een polymorfisme — die zich manifesteert wanneer één enkele nucleotide (A, C, G of T) in het genoom verschilt tussen individuen van dezelfde biologische soort. De twee DNA fragmenten AAGCCTA en AAGCTTA van verschillende individuen hebben bijvoorbeeld een andere nucleotide op de vijfde positie. We zeggen in dit geval dat er twee verschillende allelen zijn.
Deze genetische variaties zorgen ervoor dat verschillende individuen meer of minder vatbaar zijn voor bepaalde ziekten. De ernst van de ziekte en de manier waarop ons lichaam reageert op behandelingen worden ook bepaald door genetische varianties. Zo wordt de mutatie van één enkele nucleotide in het Apolipoprotein E gen bijvoorbeeld geassocieerd met een verhoogd risico op de ziekte van Alzheimer.
Opgave
In deze opgave worden DNA sequenties voorgesteld als strings die enkel bestaan uit de hoofdletters A, C, G en T, die de verschillende nucleotiden voorstellen. Gevraagd wordt:
- Schrijf een functie SNP waaraan twee DNA sequenties als argument moeten doorgegeven worden. Indien de twee sequenties even lang zijn, en slechts op één positie een verschillende nucleotide hebben, moet de functie een tuple teruggeven waarvan het eerste element de positie aangeeft waarop de twee sequenties van elkaar verschillen (posities worden geïndexeerd vanaf nul), en het tweede en derde element respectievelijk de nucleotiden aangeven die op die positie voorkomen in de eerste en tweede sequentie. Anders moet de functie de waarde None teruggeven.
- SNPs worden doorgaans gevonden door het genoom (of een lang fragment ervan) van een individu te screenen op basis van een read (een korter DNA fragment) van een ander individu. Men zegt dan dat er een SNP voorkomt wanneer op een bepaalde positie in het genoom alle overeenkomstige nucleotiden van de read teruggevonden worden, behalve op één enkele positie. Deze afwijkende positie geeft dan de positie van de SNP aan. Uiteraard is het mogelijk dat er op deze manier voor één enkele read verschillende SNPs gevonden worden in het genoom, zoals aangegeven in onderstaand voorbeeld. Gebruik de functie SNP om een functie SNPs te schrijven, waaraan de DNA sequentie van een genoom en een read moeten doorgegeven worden. De functie moet een lijst teruggeven met de posities van alle SNPs die in het genoom gevonden worden. De posities moeten in stijgende volgorde voorkomen in de lijst, en posities worden geïndexeerd vanaf 0 ten opzichte van de start van het genoom.
Voorbeeld
>>> SNP('AAGCCTA', 'AAGCTTA') (4, 'C', 'T') >>> SNP('AAGCCTAA', 'AAGCTTA') >>> SNP('AAGCTTA', 'AAGCTTA') >>> SNP('AAGCCCA', 'AAGCTTA') >>> SNPs('AGCTGATAAGCCTAAGCGCT', 'AAGCTTA') [11] >>> SNPs('ATCGTAAGCCTAAGGCTACGCTTAGAGATA', 'AAGCTTA') [9, 18] >>> SNPs('AAGCCTAAGCCTA', 'AAGCTTA') [4, 10]
| Added by: | Peter Dawyndt |
| Date: | 2012-12-19 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

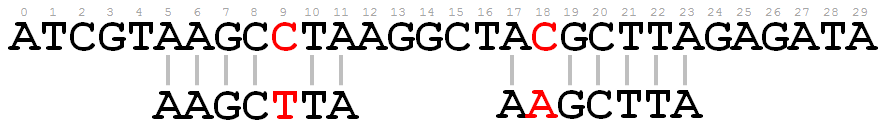
 RSS
RSS