PROG0349 - Trypsin
In this exercise we represent protein sequences as strings that only contain upper case letters. Each letter represents an amino acid within the protein sequence. Trypsin is a serine protease found in the digestive system of humans and many other vertebrates, where it helps to digest food proteins. The enzyme has a very specific function — it only cleaves peptide chains at the carboxyl side of the amino acids lysine (represented by the letter K) or arginine (represented by the letter R). As such, it is often used in laboratories studying protein structures.
High-performance liquid chromatography (HPLC) is a chromatographic technique used to separate the components in a mixture, to identify each component, and to quantify each component. When combined with shotgun tandem mass spectrometric methods, the active proteins within a biological sample may be determined. A trypsin digest is used to cleave the proteins in a sample downstream to every K or R. The individual components that result after the cleavage step are called tryptic peptides. The amino acid sequence of these tryptic peptides may then be determined by means of mass spectrometry. However, most devices have a detection limit that only allows to determine the amino acid sequence of peptides having a length between 5 and 50 amino acids. Further, if the last peptide of the protein chain does not end with K or R, it will not be picked up by the mass spectrometer.
Software suites such as Unipept are based on large protein databases, containing tryptic peptides taken from more than 29 million known proteins. This online platform can be used to determine both the diversity and the functional activity of a biological sample by comparing the tryptic peptides found in the sample with those found in the database.
Assignment
- Write a function trypsin that takes a protein sequence as its argument. The function must return the list of tryptic peptides that results from cleaving the given protein sequence by trypsin. The order of the peptides in the list should correspond to the order of the peptides in the protein sequence.
- Write a function massSpectrometer that takes a protein sequence as its argument. Analogous to the function trypsin,the function must return the list of tryptic peptides that results from cleaving the given protein sequence by trypsin. However, only those tryptic peptides that are with the detection limit of a mass spectrometer (length between 5 and 50 amino acids, including limits; ending with K or R) must be included in the list.
Example
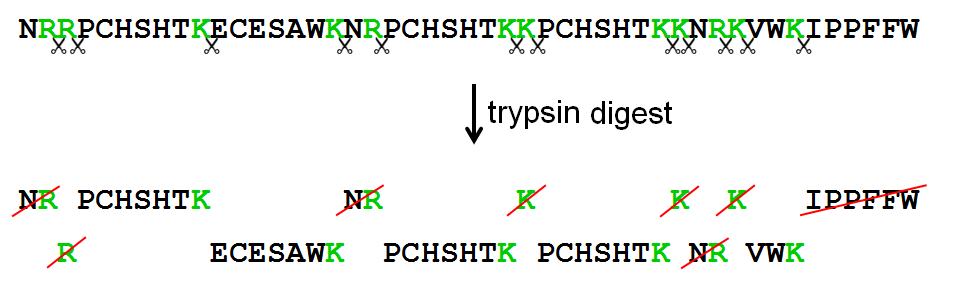
>>> trypsin('NRRPCHSHTKECESAWKNRPCHSHTKKPCHSHTKKNRKVWKIPPFFW') ['NR', 'R', 'PCHSHTK', 'ECESAWK', 'NR', 'PCHSHTK', 'K', 'PCHSHTK', 'K', 'NR', 'K', 'VWK', 'IPPFFW'] >>> trypsin('HAEWTDNQCCPVLKECESAWKYEMWQHPGEQHKRRRYEMWQHPGEQHKPCHSHTKVWKRY') ['HAEWTDNQCCPVLK', 'ECESAWK', 'YEMWQHPGEQHK', 'R', 'R', 'R', 'YEMWQHPGEQHK', 'PCHSHTK', 'VWK', 'R', 'Y'] >>> massSpectrometer('NRRPCHSHTKECESAWKNRPCHSHTKKPCHSHTKKNRKVWKIPPFFW') ['PCHSHTK', 'ECESAWK', 'PCHSHTK', 'PCHSHTK'] >>> massSpectrometer('HAEWTDNQCCPVLKECESAWKYEMWQHPGEQHKRRRYEMWQHPGEQHKPCHSHTKVWKRY') ['HAEWTDNQCCPVLK', 'ECESAWK', 'YEMWQHPGEQHK', 'YEMWQHPGEQHK', 'PCHSHTK']
References
Eiwitsequenties worden in deze opgave voorgesteld als strings die enkel hoofdletters bevatten. Elke hoofdletter stelt een aminozuur van de sequentie voor. Trypsine is een eiwitafbrekend enzym dat voedingseiwitten afbreekt in de dunne darm van de mens en verschillende diersoorten. Dit enzym heeft een zeer specifieke functie — het splitst alleen peptidebindingen waarvan de carboxylgroep afkomstig is van één van de basische aminozuren lysine (voorgesteld door de letter K) en arginine (voorgesteld door de letter R) — en wordt daarom in het laboratorium veel toegepast bij structureel onderzoek van eiwitten.
High-performance liquid chromatography (HPLC) is een scheidingstechniek die kan gecombineerd worden met shotgun tandem massaspectrometrische methoden om de actieve eiwitten in een biologisch staal te bepalen. Hierbij wordt een trypsine digest gebruikt om de eiwitten van het staal open te knippen in verschillende stukken na elke K of R in de sequentie. Deze afzonderlijke stukken worden tryptische peptiden genoemd. De sequentie van tryptische peptiden kan met een massaspectrometer bepaald worden. De meeste toestellen hebben echter een detectielimiet die enkel toelaat om peptiden met een lengte tussen 5 en 50 uit te lezen. Als de laatste peptide van de eiwitsequentie zelf niet op K of R eindigt, dan wordt ze ook niet opgepikt door de massaspectrometer.
Toepassingen zoals Unipept bouwen een grote eiwitdatabank op, die tryptische peptiden bevat van meer dan 29 miljoen gekende eiwitten. Deze toepassing kan zowel de diversiteit als de functionele activiteit van een biologisch staal onderzoeken, door na te gaan welke eiwitten corresponderen met de tryptische peptiden die uit het staal gesequeneerd worden.
Opgave
- Schrijf een functie trypsine waaraan een eiwitsequentie als argument moet doorgegeven worden. De functie moet een lijst van de tryptische peptiden teruggeven die verkregen worden door het eiwit af te breken met trypsine. De volgorde van de peptiden in de lijst moet dezelfde zijn als de volgorde van de peptiden in het eiwit.
- Schrijf een functie massaspectrometer waaraan een eiwitsequentie als argument moet doorgegeven worden. Analoog aan de functie trypsine moet ook deze functie de lijst van tryptische peptiden van het eiwit teruggeven, maar dan enkel de peptiden die binnen de detectielimiet van een massaspectrometer vallen (lengte tussen 5 en 50 aminozuren, grenzen inbegrepen; eindigen op K of R).
Voorbeeld
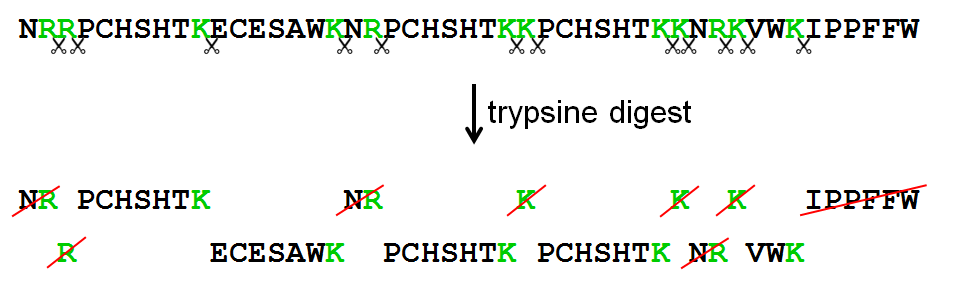
>>> trypsine('NRRPCHSHTKECESAWKNRPCHSHTKKPCHSHTKKNRKVWKIPPFFW') ['NR', 'R', 'PCHSHTK', 'ECESAWK', 'NR', 'PCHSHTK', 'K', 'PCHSHTK', 'K', 'NR', 'K', 'VWK', 'IPPFFW'] >>> trypsine('HAEWTDNQCCPVLKECESAWKYEMWQHPGEQHKRRRYEMWQHPGEQHKPCHSHTKVWKRY') ['HAEWTDNQCCPVLK', 'ECESAWK', 'YEMWQHPGEQHK', 'R', 'R', 'R', 'YEMWQHPGEQHK', 'PCHSHTK', 'VWK', 'R', 'Y'] >>> massaspectrometer('NRRPCHSHTKECESAWKNRPCHSHTKKPCHSHTKKNRKVWKIPPFFW') ['PCHSHTK', 'ECESAWK', 'PCHSHTK', 'PCHSHTK'] >>> massaspectrometer('HAEWTDNQCCPVLKECESAWKYEMWQHPGEQHKRRRYEMWQHPGEQHKPCHSHTKVWKRY') ['HAEWTDNQCCPVLK', 'ECESAWK', 'YEMWQHPGEQHK', 'YEMWQHPGEQHK', 'PCHSHTK']
Bronnen
| Added by: | Peter Dawyndt |
| Date: | 2013-03-30 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS