PROG0303 - Genome density
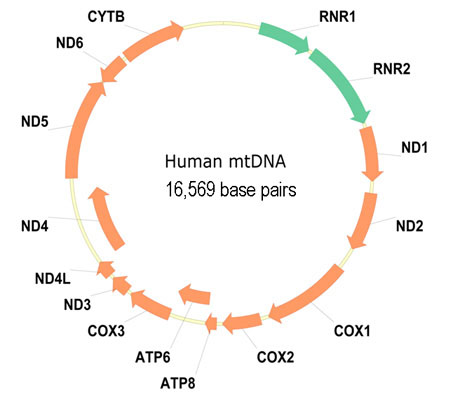
The genome of an organism is the whole of hereditary infromation in a cell. This hereditary information is either coded in DNA or — for some types of viruses — in RNA. Genes are structural components of DNA and RNA that code for a polypeptide or an RNA chain that has a certain function in an organism. The image below, for example, shows the different genes that are situated on a ring-shaped mitochondrial DNA (mrDNA) of a human being. The arrows that are used in the representation of genes, indicate that they can be oriented both forward and backward (DNA is double-stranded). A gene is localized on a genome by giving the position of the first and last base of the gene. Positions are indicated with regard to the start of the genome (for ring-shaped genomes, an initial position is chosen), that gets index 1.
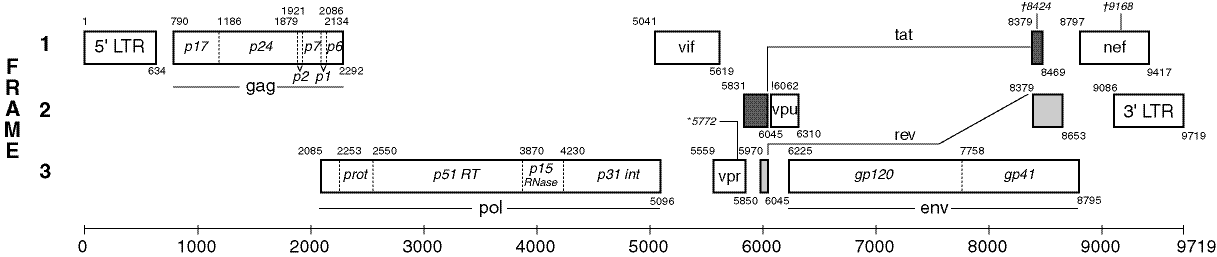
What is unclear in the image above, is that genes can also (partially) overlap. This is clear in the structure of the HIV virus (an RNA lentivirus that causes AIDS) that is given below. The genes are indicated as rectangles (RNA has only one string, which results in all genes being oriented forward).
The density of a genome is a percentage that indicates how many positions within the genome are taken by genes (or other structural elements). The genome density can be determined by summing up the lengths of the genes and then dividing it by the length of the genome. This, however, is a naive way of working, that doesn't take the overlapping genes into account and consequentially calculates some positions multiple times. A better way consists of determining what the percentage of the positions within the genome that are situated in at least one gene.
Assignment
Define a class Gene which can be used to represent gene objects that contains the following methods:
- An initializing method to which an initial position and an end position must be given as an argument. The initial position indicates the position of the first base of the gene, and the stop position indicates the position of the last base. If the stop position is smaller than the initial position, the gene is oriented backwards.
- A method __len__ that prints the length of the gene as a result.
- A method __repr__ that prints the string representation of a gene of the format "Gene(start, stop)", where start and stop respectively represent the initial and end positions of the gene.
- A method __str__ that prints the string representation of a gene of the format "start..stop" for forward genes, and of the format "complement(stop..start)" for backwards genes. Here, start and stop respectively represent the initial and end positions of the gene.
Also, define another class Genome which can be used to represent genome objects. Genome objects must be able to keep the position of their genes, and contain the following methods:
- An initializing method to which the length of the genome must be given as an argument.
- A method__len__ that prints the length of a genome as a result.
- A method addGene to which a gene object must be given as an argument. By calling this method (multiple times), the positions of the genes on the genome can be given. The method must print an AssertionError with the message invalid coordinate if the initial or end position of the given gene object are not within the boundaries of the genome.
- A method density that prints the genome density as a floating point value. This method has an optional Boolean parmeter overlap with standard value True. If the value False is given as an argument to the parameter overlap, the genome density must be calculated in the naive way, without taking into account that the genes may overlap. If the value True is given as an argument to the parameter overlap, the genome density must be calculating with the overlaps taken into account.
Example
>>> gene1 = Gene(3309, 4264) >>> len(gene1) 956 >>> gene1 Gene(3309, 4264) >>> print(gene1) 3309..4264 >>> gene2 = Gene(14675, 14151) >>> len(gene2) 525 >>> gene2 Gene(14675, 14151) >>> print(gene2) complement(14151..14675) >>> hiv = Genome(9719) >>> len(hiv) 9719 >>> hiv.addGene(Gene(1, 634)) >>> hiv.addGene(Gene(790, 2292)) >>> hiv.addGene(Gene(2085, 5096)) >>> hiv.addGene(Gene(5041, 5619)) >>> hiv.addGene(Gene(5559, 5850)) >>> hiv.addGene(Gene(5831, 6045)) >>> hiv.addGene(Gene(5970, 6045)) >>> hiv.addGene(Gene(6062, 6310)) >>> hiv.addGene(Gene(6225, 8795)) >>> hiv.addGene(Gene(8379, 8424)) >>> hiv.addGene(Gene(8379, 8653)) >>> hiv.addGene(Gene(8797, 9417)) >>> hiv.addGene(Gene(9086, 9719)) >>> hiv.density() 98.2302706039716 >>> hiv.density(overlap=False) 110.16565490276777 >>> hiv.addGene(Gene(8888, 9999)) Traceback (most recent call last): AssertionError: invalid coordinate
Het genoom van een organisme is het geheel aan erfelijke informatie in een cel. Deze erfelijke informatie zit ofwel gecodeerd in DNA of — voor sommige soorten virussen — in RNA. Genen zijn structurele componenten van DNA en RNA die coderen voor een polypeptide of voor een RNA keten die een bepaalde functie vertolkt voor het organisme. Onderstaande afbeelding toont bijvoorbeeld de verschillende genen die gelegen zijn op het ringvormig mitochondriaal DNA (mtDNA) van een mens. De pijlen die gebruikt worden bij de voorstelling van de genen, geven aan dat ze zowel voorwaarts als achterwaarts kunnen georiënteerd zijn (DNA is dubbelstrengig). Een gen wordt gelocaliseerd op een genoom door de positie van de eerste en de laatste base van het gen op te geven. Posities worden aangegeven ten opzichte van de start van het genoom (bij ringvormige genomen wordt een startpositie gekozen), die zelf index 1 krijgt.
Wat uit bovenstaande afbeelding minder duidelijk is, is dat genen elkaar ook (gedeeltelijk) kunnen overlappen. Dat is duidelijker te zien bij de structurele opbouw van het HIV virus (een RNA lentivirus dat AIDS veroorzaakt) dat hieronder wordt weergegeven. De genen worden hierbij als rechthoeken aangeduid (RNA heeft immers maar één streng, waardoor alle genen in dit geval voorwaarts georiënteerd zijn).
De dichtheid van een genoom is een percentage dat aangeeft hoeveel posities binnen het genoom bedekt zijn door genen (of andere structurele elementen). De genoomdichtheid kan bepaald worden door de lengtes van de genen te sommeren, en dit te delen door de lengte van het genoom. Dit is echter een naïeve manier van werken, die geen rekening houdt met overlappende genen en dus sommige posities meerdere keren in rekening kan brengen. Een betere manier bestaat erin te bepalen wat het percentage is van de posities binnen het genoom die binnen minstens één gen gelegen zijn.
Opgave
Definieer een klasse Gen waarmee genobjecten kunnen voorgesteld worden die over volgende methoden beschikken:
- Een initialisatiemethode waaraan een startpositie en een stoppositie als argument moeten doorgegeven worden. De startpositie geeft aan waar de eerste base van het gen gelegen is, en de stoppositie waar de laatste base gelegen is. Als de stoppositie kleiner is dan de startpositie, dan is het gen achterwaarts georiënteerd.
- Een methode __len__ die de lengte van het gen als resultaat teruggeeft.
- Een methode __repr__ die de stringvoorstelling van een gen teruggeeft onder de vorm "Gen(start, stop)", waarbij start en stop respectievelijk de start- en stopposities van het gen voorstellen.
- Een methode __str__ die de stringvoorstelling van een gen teruggeeft onder de vorm "start..stop" voor voorwaartse genen, en onder de vorm "complement(stop..start)" voor achterwaartse genen. Hierbij stellen start en stop respectievelijk de start- en stopposities van het gen voor.
Definieer ook nog een klasse Genoom waarmee genoomobjecten kunnen voorgesteld worden. Genoomobjecten moeten kunnen bijhouden waar hun genen gelegen zijn, en beschikken over volgende methoden:
- Een initialisatiemethode waaraan de lengte van het genoom als argument moet doorgegeven worden.
- Een methode __len__ die de lengte van het genoom als resultaat teruggeeft.
- Een methode genToevoegen waaraan een genobject als argument moet doorgegeven worden. Door deze methode (verschillende keren) aan te roepen, kan opgegeven worden waar de verschillende genen op het genoom gelegen zijn. De methode moet een AssertionError met de tekst ongeldige coordinaat opwerpen indien de start- of stoppositie van het gegeven genobject niet binnen de grenzen van het genoom ligt.
- Een methode dichtheid die de genoomdichtheid als een floating point waarde teruggeeft. Deze methode heeft een optionele Booleaans parameter overlap met als standaardwaarde True. Als de waarde False als argument wordt doorgegeven aan de parameter overlap, dan moet de genoomdichtheid op de naïeve manier berekend worden, zonder rekening te houden met mogelijke overlappende genen. Als de waarde True als argument wordt doorgegeven aan de parameter overlap, dan moet de genoomdichtheid berekend worden rekening houdend met mogelijke overlappende genen.
Voorbeeld
>>> gen1 = Gen(3309, 4264) >>> len(gen1) 956 >>> gen1 Gen(3309, 4264) >>> print(gen1) 3309..4264 >>> gen2 = Gen(14675, 14151) >>> len(gen2) 525 >>> gen2 Gen(14675, 14151) >>> print(gen2) complement(14151..14675) >>> hiv = Genoom(9719) >>> len(hiv) 9719 >>> hiv.genToevoegen(Gen(1, 634)) >>> hiv.genToevoegen(Gen(790, 2292)) >>> hiv.genToevoegen(Gen(2085, 5096)) >>> hiv.genToevoegen(Gen(5041, 5619)) >>> hiv.genToevoegen(Gen(5559, 5850)) >>> hiv.genToevoegen(Gen(5831, 6045)) >>> hiv.genToevoegen(Gen(5970, 6045)) >>> hiv.genToevoegen(Gen(6062, 6310)) >>> hiv.genToevoegen(Gen(6225, 8795)) >>> hiv.genToevoegen(Gen(8379, 8424)) >>> hiv.genToevoegen(Gen(8379, 8653)) >>> hiv.genToevoegen(Gen(8797, 9417)) >>> hiv.genToevoegen(Gen(9086, 9719)) >>> hiv.dichtheid() 98.2302706039716 >>> hiv.dichtheid(overlap=False) 110.16565490276777 >>> hiv.genToevoegen(Gen(8888, 9999)) Traceback (most recent call last): AssertionError: ongeldige coordinaat
| Added by: | Peter Dawyndt |
| Date: | 2012-12-30 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS