| Submit | All submissions | Best solutions | Back to list |
PROG0038 - The billion-year war |
The war between viruses and bacteria has been waged for over a billion years. Viruses called bacteriophages (or simply phages) require a bacterial host to propagate, and so they must somehow infiltrate the bacterium. Such deception can only be achieved if the phage understands the genetic framework underlying the bacterium's cellular functions. The phage's goal is to insert DNA that will be replicated within the bacterium and lead to the reproduction of as many copies of the phage as possible, which sometimes also involves the bacterium's demise.
To defend itself, the bacterium must either obfuscate its cellular functions so that the phage cannot infiltrate it, or better yet, go on the counterattack by calling in the air force. Specifically, the bacterium employs aerial scouts called restriction enzymes, which operate by cutting through viral DNA to cripple the phage. But what kind of DNA are restriction enzymes looking for?
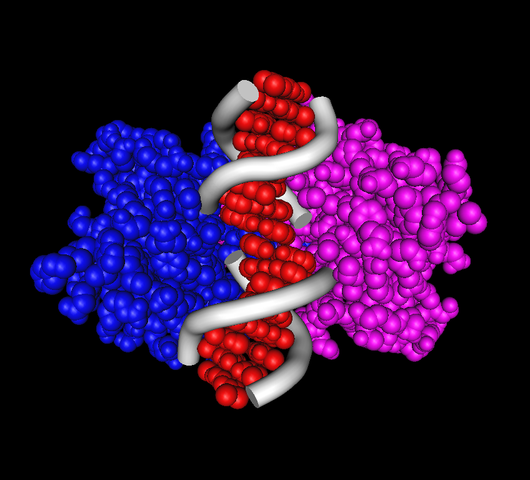
The restriction enzyme is a homodimer, which means that it is composed of two identical substructures. Each of these structures separates from the restriction enzyme in order to bind to and cut one strand of the phage DNA molecule. Both substructures are pre-programmed with the same target string containing 4 to 12 nucleotides to search for within the phage DNA (see figure above). The chance that both strands of phage DNA will be cut (thus crippling the phage) is greater if the target is located on both strands of phage DNA, as close to each other as possible. By extension, the best chance of disarming the phage occurs when the two target copies appear directly across from each other along the phage DNA, a phenomenon that occurs precisely when the target is equal to its own reverse complement. Eons of evolution have made sure that most restriction enzyme targets now have this form.
Assignment
In this assignment we represent a DNA sequence as a string that only contains the uppercase letters A, C, G and T. The reverse complement of formed by reversing the string and taking the complement of each character. The characters A and T are complement each other, and so do the characters C and G. We must also reverse the string in addition to taking complements because of the directionality of DNA: DNA replication and transcription occurs from the 3' end to the 5' end, and the 3' end of one strand is opposite from the 5' end of the complementary strand. Thus, if we were to simply take complements, then we would be reading the second strand in the wrong direction.
A DNA sequence is a reverse palindrome if it is equal to its reverse complement. For instance, GCATGC is a reverse palindrome because its reverse complement is GCATGC (see figure above). Your task:
- Write a function reverseComplement that takes a DNA sequence. The function must return the reverse complement of the given DNA sequence.
- Write a function reversePalindrome that takes a DNA sequence. The function must return a Boolean value that indicates whether or not the given DNA sequence is a reverse palindrome.
- Write a function restrictionSites that takes a DNA sequence. The function must return a list containing all restriction sites in the given DNA sequence. A restriction site is a position in a DNA sequence where a reverse palindrome is located. Each restriction site is represented by a tuple that contains the position of the first letter of the palindrome, together with the palindrome itself. Here we assume that the first character of the DNA sequence is at position 1, the second letter at position 2, and so on. The restriction sites must be sorted, first according to increasing start position and then according to increasing length of the palindromes. The function has two additional optional arguments minLength (default value: 4) and maxLength (default value: 12) that respectively take the minimal and maximal length of the palindromes that must be taken into account to determine the restriction sites.
Example
>>> reverseComplement('GATATC') 'GATATC' >>> reverseComplement('GCATGC') 'GCATGC' >>> reverseComplement('AGCTTC') 'GAAGCT' >>> reversePalindrome('GATATC') True >>> reversePalindrome('GCATGC') True >>> reversePalindrome('AGCTTC') False >>> restrictionSites('TCAATGCATGCGGGTCTATATGCAT') [(4, 'ATGCAT'), (5, 'TGCA'), (6, 'GCATGC'), (7, 'CATG'), (17, 'TATA'), (18, 'ATAT'), (20, 'ATGCAT'), (21, 'TGCA')] >>> restrictionSites('AAGTCATAGCTATCGATCAGATCAC', minLength=5) [(6, 'ATAGCTAT'), (7, 'TAGCTA'), (12, 'ATCGAT')] >>> restrictionSites('ATATTCAGTCATCGATCAGCTAGCA', maxLength=5) [(1, 'ATAT'), (12, 'TCGA'), (14, 'GATC'), (18, 'AGCT'), (20, 'CTAG')]
Epilogue
You may be curious how the bacterium prevents its own DNA from being cut by restriction enzymes. The short answer is that it locks itself from being cut through a chemical process called DNA methylation. DNA methylation is a chemical process that a cell applies to its own DNA by bonding methyl groups ($CH_3$) to nucleotides, which effectively locks them from being involved in a reaction (especially those involving further bonding, like transcription).
Methylation serves a number of fascinating practical purposes. In one example, restriction enzymes employed by a bacterium would not be capable of discriminating between the foreign DNA of a phage and the bacterium's own DNA, so the bacterium methylates its DNA to protect it from its own restriction enzymes.
Methylation is also a remarkable way to regulate gene activity, as methylated DNA can be inherited, which has opened up a brand new field called epigenetics. This field studies functionally relevant modifications to the genome that do not involve a change in the genome's sequence of nucleotides. In short, the ultimate truth is that there is a lot more to inheritance than simply replicating DNA!
Methylation usually occurs at CpG sites, where cytosine and guanine nucleotides appear consecutively. In recent years, researchers have shown that DNA methylation occurs in higher organisms and that it is important for normal development: methylated areas of the genome are protected from transcription activators and remain inactive. These "silent" parts of the genome are called heterochromatin.
De oorlog tussen virussen en bacteriën is al meer dan een miljard jaar aan de gang. De aanleiding voor deze oorlog is dat een bepaald soort virus — bacteriofagen (of kortweg fagen) genaamd — een bacteriële gastheer nodig heeft om zich te kunnen voortplanten en daarvoor ongemerkt de bacteriële cellen moet zien binnen te dringen. Een dergelijke infiltratie kan echter alleen maar operationeel gebracht worden als de faag doorheeft hoe de genetische mechaniek in elkaar zit die bepalend is voor de cellulaire functies van de bacterie. Het enige doel van de faag is immers om zijn eigen DNA binnen te smokkelen in het bacteriële DNA, zodat het mee gekopieerd wordt met de bacterie en resulteert in de reproductie van zoveel mogelijk kopieën van de faag. Ook al heeft dit soms finaal de dood van de bacteriële cel tot gevolg.
Om zichzelf te beschermen, moet een bacteriële cel ofwel haar cellulaire functies camoufleren zodat de faag niet kan binnendringen, of beter nog, kan ze in de tegenaanval gaan door versterking in te roepen van de luchtmacht. Meer specifiek maakt de bacterie gebruik van een soort drones die restrictie-enzymen genoemd worden. Deze herkennen viraal DNA en knippen het in stukken om zo de faag te verminken. Maar naar welk soort DNA zijn de restrictie-enzymen nu specifiek op zoek?
Het restrictie-enzym is een homodimeer, wat betekent dat het samengesteld is uit twee identieke substructuren. Om zijn werk te kunnen doen, splitst het restrictie-enzym zich op in deze twee substructuren die zich elk binden aan één streng van het DNA-molecuul van de faag, en die daarna de streng doormidden knippen. Beide substructuren zijn voorgeprogrammeerd om hetzelfde fragment van 4 tot 12 nucleotiden op te sporen in het DNA van de faag (zie bovenstaande figuur). De kans dat beide strengen van het faag-DNA zullen doorgeknipt worden om de faag voldoende te verminken, vergroot naarmate de doelfragmenten op beide strengen van het faag-DNA dichter bij elkaar liggen. Bij uitbreiding is de kans om de faag te ontwapenen het grootst als beide doelfragmenten recht tegenover elkaar liggen langs het faag-DNA. Een fenomeen dat optreedt wanneer het doelfragment precies gelijk is aan zijn omgekeerd complement (zie onderstaande figuur). Eeuwen van evolutie hebben er uiteindelijk toe geleid dat de meeste restrictie-enzymen nu doelfragmenten van deze vorm hebben.
Opgave
In deze opgave stellen we een DNA-sequentie voor als een string die enkel bestaat uit de hoofdletters A, C, G en T. Het omgekeerd complement van een DNA-sequentie wordt bekomen door de string om te keren en daarna elk karakter te vervangen door zijn complement. Hierbij zijn A en T elkaars complement, en zijn ook C en G elkaars complement. Naast het bepalen van het complement moeten we de string ook omkeren vanwege de gerichtheid van DNA: DNA-replicatie en transcriptie gebeuren vanaf het 3' uiteinde naar het 5' uiteinde, en het 3' uiteinde van de ene streng ligt tegenover het 5' uiteinde van de complementaire streng. Als we dus enkel het complement zouden nemen, dan zou de complentaire streng in de verkeerde richting gelezen worden.
We zeggen dat een DNA-sequentie een omgekeerd palindroom is, als de DNA-sequentie gelijk is aan zijn omgekeerd complement. Uit bovenstaande figuur leiden we bijvoorbeeld af dat GATATC een omgekeerd palindroom is, omdat zijn omgekeerd complement gelijk is aan GATATC. Gevraagd wordt:
- Schrijf een functie omgekeerdComplement waaraan een DNA-sequentie moet doorgegeven worden. De functie moet het omgekeerd complement van de gegeven DNA-sequentie teruggeven.
- Schrijf een functie omgekeerdPalindroom waaraan een DNA-sequentie moet doorgegeven worden. De functie moet een Booleaanse waarde teruggeven die aangeeft of de gegeven DNA-sequentie al dan niet een omgekeerd palindroom is.
- Schrijf een functie restrictieplaatsen waaraan een DNA-sequentie moet doorgegeven worden. De functie moet een lijst met alle restrictieplaatsen in de gegeven DNA-sequentie teruggeven. Een restrictieplaats is een positie in een DNA-sequentie waar een omgekeerd palindroom gevonden wordt. Elke restrictieplaats wordt voorgesteld door een tuple dat de positie van de eerste letter van het palindroom bevat, samen met het palindroom zelf. Hierbij staat de eerste letter van de DNA-sequentie op positie 1, de tweede letter op positie 2, enzoverder. De restrictieplaatsen moeten gesorteerd worden, eerst volgens oplopende startpositie en daarna volgens oplopende lengte van de palindromen. De functie heeft ook nog twee optionele parameters minLengte (standaardwaarde: 4) en maxLengte (standaardwaarde: 12) waaraan respectievelijk de minimale en maximale lengte kunnen doorgegeven worden van de palindromen die in aanmerking genomen worden om de restrictieplaatsen te bepalen.
Voorbeeld
>>> omgekeerdComplement('GATATC') 'GATATC' >>> omgekeerdComplement('GCATGC') 'GCATGC' >>> omgekeerdComplement('AGCTTC') 'GAAGCT' >>> omgekeerdPalindroom('GATATC') True >>> omgekeerdPalindroom('GCATGC') True >>> omgekeerdPalindroom('AGCTTC') False >>> restrictieplaatsen('TCAATGCATGCGGGTCTATATGCAT') [(4, 'ATGCAT'), (5, 'TGCA'), (6, 'GCATGC'), (7, 'CATG'), (17, 'TATA'), (18, 'ATAT'), (20, 'ATGCAT'), (21, 'TGCA')] >>> restrictieplaatsen('AAGTCATAGCTATCGATCAGATCAC', minLengte=5) [(6, 'ATAGCTAT'), (7, 'TAGCTA'), (12, 'ATCGAT')] >>> restrictieplaatsen('ATATTCAGTCATCGATCAGCTAGCA', maxLengte=5) [(1, 'ATAT'), (12, 'TCGA'), (14, 'GATC'), (18, 'AGCT'), (20, 'CTAG')]
Epiloog
Bij het lezen van de inleiding heb je je misschien afgevraagd hoe bacteriën zichzelf beschermen en vermijden dat de restrictie-enzymen hun eigen DNA doorknippen. Het korte antwoord op deze vraag is dat bacteriële cellen hun eigen DNA afschermen om doorgeknipt te worden door een chemisch proces dat DNA-methylatie genoemd wordt. Bij DNA-methylatie bindt een cel methylgroepen ($CH_3$) aan sommige nucleotiden, waardoor bepaalde chemische reacties afgeblokt worden (in het bijzonder andere bindingsreacties zoals transcriptie).
Methylatie heeft een aantal bijzonder nuttige toepassingen. Een voorbeeld hiervan is dat de restrictie-enzymen die door bacteriën gebruikt worden normaalgezien geen onderscheid kunnen maken tussen vreemd DNA dat afkomstig is van een faag en het eigen DNA van de bacterie. Daarom methyleert de bacterie haar eigen DNA om het zo te beschermen tegen haar eigen restrictie-enzymen.
Bovendien biedt methylatie ook een opmerkelijke manier om de activiteit van genen te regelen, omdat gemethyleerd DNA kan doorgegeven worden aan het nageslacht. Dit inzicht was de start van een volledig nieuw onderzoeksdomein dat epigenetica genoemd wordt. Hierbij worden functioneel relevante modificaties van het genoom onderzocht die geen wijzigen aan de nucleotiden van het genoom met zich meebrengen. Kortom, de ultieme waarheid is dat erfelijkheid veel meer inhoudt dan het eenvoudigweg repliceren van DNA!
Methylatie doet zich meestal voor op CpG plaatsen, waar cytosine en guanine na elkaar voorkomen. Recent onderzoek heeft aangetoond dat DNA-methylatie ook voorkomt bij hogere organismen en dat het belangrijk is voor hun normale ontwikkeling: gemethyleerde gebieden van het genoom worden beschermd tegen transcriptionele activatoren en blijven daardoor inactief. Deze "stille" gebieden van het genoom worden heterochromatine genoemd.
| Added by: | Peter Dawyndt |
| Date: | 2011-07-23 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |



 RSS
RSS