| Submit | All submissions | Best solutions | Back to list |
PROG0305 - Sanger sequencing |
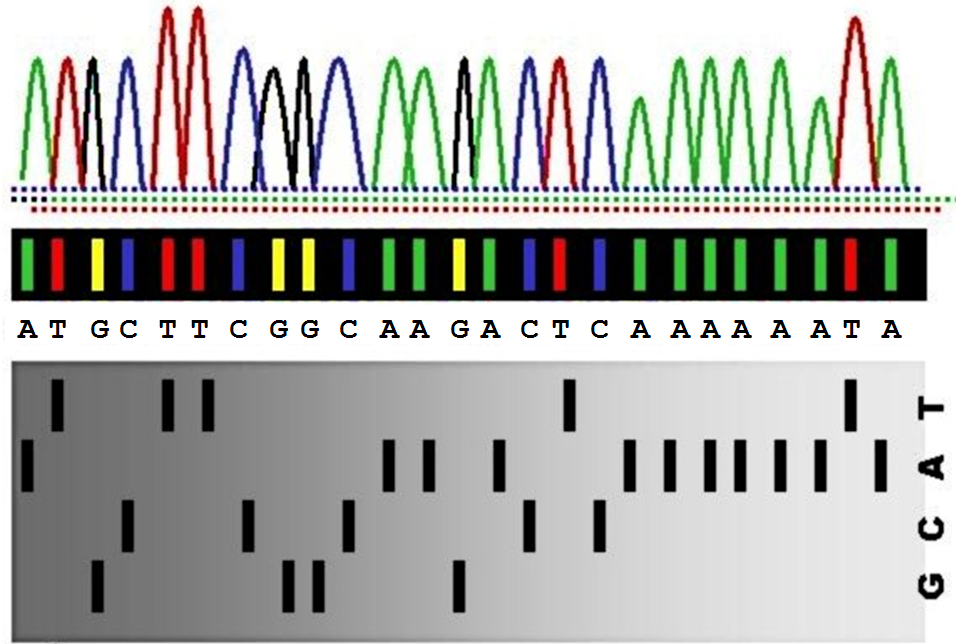
Sanger sequencing is a method used to determine the DNA sequence of an organism. Developed by Frederick Sanger and colleagues in 1977, it was the most widely used sequencing method for approximately 25 years. The method is applied by radioactively or fluorescently labelling the four DNA-bases (represented by the letters A, C, G and T), so that successive bases can be read out sequentially. The radioactive or fluorescent labels can be read automatically using 4 different detectors that determine for each position in the DNA whether a radioactive or fluorescent signal is present for each of the four bases. This results in four separate signals that are then merged into a single signal, from which the DNA sequence itself may be determined.
If the detector for base X (with X being one of the letters A, C, G or T) registers a positive signal at a certain position, the position is marked by the letter X. If no signal is detected, the position is marked by a hyphen. As such, the output of all four detectors — as measured for the DNA sequence ATGCTTCGGCAAGACTCAAAAAATA — is represented in the following format:
1111111111222222
1234567890123456789012345
detector A: A---------AA-A---AAAAAA-A
detector C: ---C--C--C----C-C--------
detector G: --G----GG---G------------
detector T: -T--TT---------T-------T-
=========================
DNA sequence: ATGCTTCGGCAAGACTCAAAAAATA
Assignment
Write a function sanger that takes a string argument that only contains the upper case letters A, C, G and T. This argument represents a DNA sequence. The function must return a string that represents the given DNA-sequence using the following format:
- In the centre are 4 lines representing the individual signals as measures by the detector for each of the four bases A, C, G and T. These lines only contain hyphens and the base letter at the positions where a signal was measured by the detector.
- Below these 4 lines is a single line that contains the same number of equality signs (=) as there are bases in the given DNA sequence.
- The given DNA-sequence appears at the bottom of the format.
- On top of the lines containing the individual signals are a number of lines that index the bases in the DNA-sequence. Indexing of the positions starts at 1. First there is a line containing the units of the indexes. On top of this line there is a line containing the tens, again topped by a (third) line containing the hundreds, and so on. If the length of the sequence is smaller than ten, no tens need to be represented and thus only a single line containing index digits must be included (analogous for sequences shorter than 100, 1000, …). Any position that has no digit for the tens (leading zeros are ignored) is filled with a space on the line representing the tens of the position indexes (analogous for the hundreds, thousands, ...).
Make sure that the last line returned by the function sanger, does not end with a newline. Take a look at the interactive Pyton session below to see some examples on how the formatting of the DNA-sequences must be done.
Example
>>> print(sanger('ATGCTTCGG')) 123456789 A-------- ---C--C-- --G----GG -T--TT--- ========= ATGCTTCGG >>> print(sanger('ATGCTTCGGCAAGACTCAAAAAATA')) 1111111111222222 1234567890123456789012345 A---------AA-A---AAAAAA-A ---C--C--C----C-C-------- --G----GG---G------------ -T--TT---------T-------T- ========================= ATGCTTCGGCAAGACTCAAAAAATA
Sangersequenering is een methode die gebruikt wordt om het DNA van een organisme te bepalen. De methode werd in 1977 ontwikkeld door Frederick Sanger en zijn collega's, en was gedurende 25 jaar de meest gebruikte sequeneringsmethode. Bij de Sangermethode worden de vier basen waaruit het DNA is opgebouwd (voorgesteld door de letters A, C, G en T) radioactief of fluorescent gelabeld, zodat opeenvolgende basen achtereenvolgens kunnen uitgelezen worden. Dit uitlezen gebeurt aan de hand van vier afzonderlijke detectoren, die voor elke positie nagaan of er een radioactief of fluorescent signaal gemeten wordt voor één van de vier mogelijke basen. Dit levert dus vier afzonderlijke signalen op die daarna kunnen samengevoegd worden tot één enkel signaal, waaruit de eigenlijke DNA sequentie kan samengesteld worden.
Als de detector voor base X (waarbij X staat voor één van de letters A, C, G of T) op een bepaalde positie een signaal heeft opgepikt, dan wordt dit voorgesteld aan de hand van de letter X. Als er geen signaal werd opgepikt, dan wordt dit voorgesteld met een koppelteken (-). Op die manier worden de signalen van de vier afzonderlijke detectoren — zoals die gemeten worden voor de DNA sequentie ATGCTTCGGCAAGACTCAAAAAATA — als volgt voorgesteld:
1111111111222222
1234567890123456789012345
detector A: A---------AA-A---AAAAAA-A
detector C: ---C--C--C----C-C--------
detector G: --G----GG---G------------
detector T: -T--TT---------T-------T-
=========================
DNA sequentie: ATGCTTCGGCAAGACTCAAAAAATA
Opgave
Schrijf een functie sanger waaraan een string als argument moet doorgegeven worden die enkel bestaat uit de hoofdletters A, C, G en T. Dit stringargument stelt een DNA sequentie voor. De functie moet als resultaat een string teruggeven waarin de gegeven DNA sequentie op de volgende manier wordt opgemaakt:
- Centraal staan vier regels die de afzonderlijke signalen weergeven zoals die worden opgepikt door de detectoren voor de basen A, C, G en T. Deze regels bevatten dus enkel koppeltekens en baseletters op de posities waar een signaal werd opgepikt.
- Onder deze vier regels staat een regel die evenveel gelijkheidstekens (=) bevat als er basen zijn in de gegeven DNA sequentie.
- Helemaal onderaan staat de gegeven DNA sequentie zelf.
- Boven de regels met de afzonderlijke signalen staan er nog een aantal regels die de posities van de basen binnen de DNA sequentie omschrijven. Posities worden hierbij geïndexeerd vanaf 1. Eerst komt een regel met de cijfers van de eenheden, daarboven een regel met de cijfers van de tientallen, daarboven een regel met de cijfers van de honderdtallen, enzoverder. Als er bijvoorbeeld geen tientallen moeten weergegeven worden, dan moet daarvoor ook geen regel uitgeschreven worden (analoog voor de honderdtallen, duizendtallen, …). Voor een positie die geen cijfer heeft voor de tientallen (voorloopnullen worden niet uitgeschreven) moet een spatie uitgeschreven worden op de regel voor de tientallen (analoog voor de honderdtallen, duizendtallen, …).
De laatste regel van de string die door de functie sanger teruggegeven wordt, mag niet eindigen op een newline. Bekijk onderstaande interactieve Python sessie voor enkele voorbeelden van hoe de opmaak moet gebeuren.
Voorbeeld
>>> print(sanger('ATGCTTCGG')) 123456789 A-------- ---C--C-- --G----GG -T--TT--- ========= ATGCTTCGG >>> print(sanger('ATGCTTCGGCAAGACTCAAAAAATA')) 1111111111222222 1234567890123456789012345 A---------AA-A---AAAAAA-A ---C--C--C----C-C-------- --G----GG---G------------ -T--TT---------T-------T- ========================= ATGCTTCGGCAAGACTCAAAAAATA
| Added by: | Peter Dawyndt |
| Date: | 2013-01-02 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS