| Submit | All submissions | Best solutions | Back to list |
PROG0306 - Tryptic peptides |
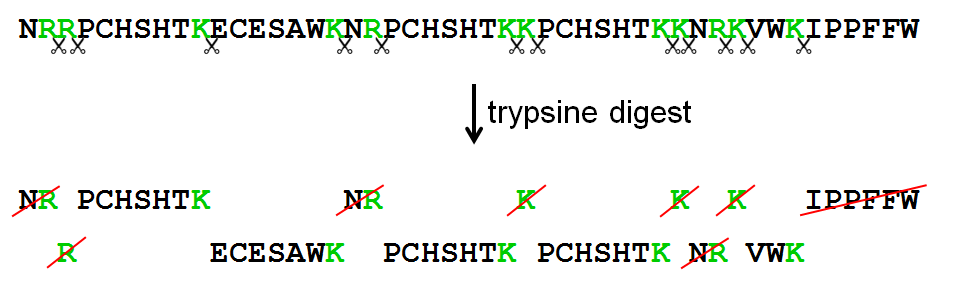
In this exercise we represent protein sequences as strings that only contain upper case letters. Each letter represents an amino acid within the protein sequence. Trypsin is a serine protease found in the digestive system of humans and many other vertebrates, where it helps to digest food proteins. The enzyme has a very specific function — it only cleaves peptide chains at the carboxyl side of the amino acids lysine (represented by the letter K) or arginine (represented by the letter R). As such, it is often used in laboratories studying protein structures.
High-performance liquid chromatography (HPLC) is a chromatographic technique used to separate the components in a mixture, to identify each component, and to quantify each component. When combined with shotgun tandem mass spectrometric methods, the active proteins within a biological sample may be determined. A trypsin digest is used to cleave the proteins in a sample downstream to every K or R. The individual components that result after the cleavage step are called tryptic peptides. The amino acid sequence of these tryptic peptides may then be determined by means of mass spectrometry. However, most devices have a detection limit that only allows to determine the amino acid sequence of peptides having a length between 5 and 50 amino acids. Further, if the last peptide of the protein chain does not end with K or R, it will not be picked up by the mass spectrometer.
Software suites such as Unipept are based on large protein databases, containing tryptic peptides taken from more than 29 million known proteins. This online platform can be used to determine both the diversity and the functional activity of a biological sample by comparing the tryptic peptides found in the sample with those found in the database.
Assignment
Define a class proteinDB that can be used to create simple protein databases. These protein databases can be used to look for proteins that contain a given list of tryptic peptides. The objects of the class proteinDB must support at least the following methods:
- An initialization method that assures that each newly created object of the class proteinDB has a property peptides that refers to a dictionary. This dictionary should initially be empty for newly created objects, but gradually it will be filled with strings as keys and sets of strings as values.
- A method addPeptide that can be used to add a new tryptic peptide to the database. The method takes two arguments: a string containing the label of a protein sequence and a string containing a tryptic peptide. Only tryptic peptides having a length between 5 and 50 residues (including boundaries) may be added to the database. In addition, tryptic peptides should end with K or R, and no other K or R may occur within the sequence. The method should throw an AssertionError with the message invalid peptide if a peptide is passed that does not meet all these conditions. Adding a tryptic peptide to the database is done by using the given tryptic peptide as a key in the dictionary that is referred to by the property peptides, and adding the given label of the protein sequences to the set that is mapped to this key by the dictionary. If the dictionary has no key for the given tryptic peptide, a new key-value pair must be added to the dictionary, with the peptide as the key and as its corresponding value a set that only contains the given label.
- A method addProtein that can be used to add all tryptic peptides of a given protein that have a length between 5 and 50 (including boundaries) to the database. Two arguments must be passed to this method: a string containing the label of the protein sequence and a string containing the protein sequence itself. The method must perform an in silico tryptic digest to cut the protein sequences into its tryptic peptides, and add each of these tryptic peptides to the database under the label of the protein sequence.
- A method addProteins that takes the location of a text file as an argument. Each line of this text file must contain the label of a protein sequence and the protein sequence itself, separated by a tab. The method must add all peptides of all proteins to the database.
- A method identify that can be used to identify proteins from the database. This method takes a collection object (e.g. a list, tuple, set, …) that contains a number of peptides. The method must return an alphabetically ordered list of the labels of all proteins from the database that contain each of the given peptides at least once.
Take care in implementing these methods that you make optimal reuse of the methods that have already been implemented.
Example
The the following interactive session we assume that the text file proteins.txt is located in the current directory.
>>> unipept = proteinDB() >>> unipept.addPeptide('PROT0001', 'ECESAWK') >>> unipept.peptides {'ECESAWK': {'PROT0001'}} >>> unipept.addPeptide('PROT0002', 'WHK') Traceback (most recent call last): AssertionError: invalid peptide >>> unipept.addPeptide('PROT0002', 'ESHLSTLAVQENEIG') Traceback (most recent call last): AssertionError: invalid peptide >>> unipept.addPeptide('PROT0002', 'NWAQNAKIGGADWDCVCR') Traceback (most recent call last): AssertionError: invalid peptide >>> unipept.addProtein('PROT0002', 'HAEWTDNQCCPVLKECESAWKYEMWQHPGEQHKRRRYEMWQHPGEQHKPCHSHTKVWKRY') >>> unipept.peptides {'ECESAWK': {'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'YEMWQHPGEQHK': {'PROT0002'}} >>> unipept.addProtein('PROT0003', 'NRRPCHSHTKECESAWKNRPCHSHTKKPCHSHTKKNRKVWKIPPFFW') >>> unipept.peptides {'ECESAWK': {'PROT0003', 'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'YEMWQHPGEQHK': {'PROT0002'}} >>> unipept.addProtein('PROT0004', 'YEMWQHPGEQHKECESAWKVPYCGFITRPCHSHTKECESAWK') >>> unipept.peptides {'ECESAWK': {'PROT0004', 'PROT0003', 'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0004', 'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'VPYCGFITR': {'PROT0004'}, 'YEMWQHPGEQHK': {'PROT0004', 'PROT0002'}} >>> unipept.identify(['VPYCGFITR']) ['PROT0004'] >>> unipept.identify({'ECESAWK', 'PCHSHTK'}) ['PROT0002', 'PROT0003', 'PROT0004'] >>> unipept.identify(('YEMWQHPGEQHK', 'ECESAWK', 'PCHSHTK')) ['PROT0002', 'PROT0004'] >>> unipept.identify({'PCHSHTK', 'VPYCGFITR'}) ['PROT0004'] >>> unipept.addProteins('proteins.txt') >>> unipept.peptides {'ECESAWK': {'PROT0005', 'PROT0004', 'PROT0003', 'PROT0002', 'PROT0001'}, 'VCEFPWFPMLINDVCR': {'PROT0007'}, 'VPYCGFITR': {'PROT0005', 'PROT0004'}, 'YEMWQHPGEQHK': {'PROT0006', 'PROT0005', 'PROT0004', 'PROT0002'}, 'PCHSHTK': {'PROT0006', 'PROT0005', 'PROT0004', 'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'CSFHCLEK': {'PROT0006'}, 'AFNYMMPNTK': {'PROT0006'}, 'AYDDEVASFPGCMMATK': {'PROT0007', 'PROT0006'}, 'FIPYYPIYSR': {'PROT0006'}, 'TLCHETMR': {'PROT0005'}, 'HTPNYGVMWMFMNEWMSYDR': {'PROT0006', 'PROT0005'}, 'CDQMHVFDIYMIAIACSWGGPPSLTK': {'PROT0007'}, 'FGHSMTR': {'PROT0005'}} >>> unipept.identify(('YEMWQHPGEQHK', 'VPYCGFITR', 'ECESAWK')) ['PROT0004', 'PROT0005'] >>> unipept.identify(('PCHSHTK', 'AYDDEVASFPGCMMATK')) ['PROT0006'] >>> unipept.identify(['NEGNLNVMK']) []
Bronnen
Eiwitsequenties worden in deze opgave voorgesteld als strings die enkel hoofdletters bevatten. Elke hoofdletter stelt een aminozuur van de sequentie voor. Trypsine is een eiwitafbrekend enzym dat voedingseiwitten afbreekt in de dunne darm van de mens en verschillende diersoorten. Dit enzym heeft een zeer specifieke functie — het splitst alleen peptidebindingen waarvan de carboxylgroep afkomstig is van één van de basische aminozuren lysine (voorgesteld door de letter K) en arginine (voorgesteld door de letter R) — en wordt daarom in het laboratorium veel toegepast bij structureel onderzoek van eiwitten.
High-performance liquid chromatography (HPLC) is een scheidingstechniek die kan gecombineerd worden met shotgun tandem massaspectrometrische methoden om de actieve eiwitten in een biologisch staal te bepalen. Hierbij wordt een trypsine digest gebruikt om de eiwitten van het staal open te knippen in verschillende stukken na elke K of R in de sequentie. Deze afzonderlijke stukken worden tryptische peptiden genoemd. De sequentie van tryptische peptiden kan met een massaspectrometer bepaald worden. De meeste toestellen hebben echter een detectielimiet die enkel toelaat om peptiden met een lengte tussen 5 en 50 uit te lezen. Als de laatste peptide van de eiwitsequentie zelf niet op K of R eindigt, dan wordt ze ook niet opgepikt door de massaspectrometer.
Toepassingen zoals Unipept bouwen een grote eiwitdatabank op, die tryptische peptiden bevat van meer dan 29 miljoen gekende eiwitten. Deze toepassing kan zowel de diversiteit als de functionele activiteit van een biologisch staal onderzoeken, door na te gaan welke eiwitten corresponderen met de tryptische peptiden die uit het staal gesequeneerd worden.
Opgave
Definieer een klasse eiwitDB waarmee eenvoudige eiwitdatabanken kunnen aangemaakt worden. In deze eiwitdatabanken kan dan gezocht worden naar eiwitten die een opgegeven lijst van tryptische peptiden bevatten. De objecten van de klasse eiwitDB moeten de volgende methoden hebben:
- Een initialisatiemethode die ervoor zorgt dat elk nieuw aangemaakt object van de klasse eiwitDB een eigenschap peptiden heeft die verwijst naar een dictionary. Bij een nieuw aangemaakt object is deze dictionary nog leeg, maar gaandeweg kan deze dictionary opgevuld worden met strings als sleutels en verzamelingen van strings als waarden.
- Een methode peptideToevoegen die kan gebruikt worden om een nieuwe tryptische peptide aan de databank toe te voegen. Aan deze methode moeten twee argumenten doorgegeven worden: een string die het label van een eiwitsequentie bevat, en een string die een tryptische peptide bevat. Enkel tryptische peptiden met lengte tussen 5 en 50 (grenzen inbegrepen) mogen aan de databank toegevoegd worden. Bovendien moet een tryptische peptide eindigen op K of R, en mag elders in de sequentie geen K of R voorkomen. De methode moet een AssertionError met de tekst ongeldige peptide opwerpen als er een peptide wordt doorgegeven die niet aan deze voorwaarden voldoet. Het toevoegen van een tryptische peptide aan de databank gebeurt door in de dictionary waarnaar de eigenschap peptiden verwijst, het label van de eiwitsequentie toe te voegen aan de verzameling die correspondeert met de sleutel die gelijk is aan de peptide die aan de methode werd doorgegeven. Als de dictionary nog geen sleutel heeft voor de gegeven peptide, dan moet er in de dictionary een nieuw sleutel-waarde paar toegevoegd worden, met de peptide als sleutel en als waarde een verzameling die enkel het gegeven label bevat.
- Een methode eiwitToevoegen die kan gebruikt worden om alle tryptische peptiden met lengte tussen 5 en 50 (grenzen inbegrepen) van een gegeven eiwitsequentie aan de databank toe te voegen. Aan deze methode moeten twee argumenten doorgegeven worden: een string die het label van de eiwitsequentie bevat, en een string met de eiwitsequentie zelf. De methode moet de eiwitsequentie opbreken in tryptische peptiden, en elk van deze peptiden aan de databank toevoegen onder het label van de eiwitsequentie.
- Een methode eiwittenToevoegen waaraan een bestandsnaam als argument moet doorgegeven worden. Deze bestandsnaam moet verwijzen naar een tekstbestand, waarvan elke regel een label en een eiwitsequentie bevat, van elkaar gescheiden door een tab. Deze methode moet alle peptiden van alle eiwitten uit het bestand toevoegen aan de databank.
- Een methode identificeer die kan gebruikt worden om eiwitten uit de databank te identificeren. Aan deze methode moet een collectieobject (dat kan dus een lijst, tuple, verzameling, … zijn) doorgegeven worden, waarin een aantal peptiden vervat zitten. De methode moet een alfabetisch gesorteerde lijst teruggeven met de labels van alle eiwitten uit de databank waarin elk van de gegeven peptiden minstens één keer voorkomt.
Zorg er bij de implementatie van al deze methoden voor dat je optimaal gebruik maakt van de methoden die je reeds eerder geïmplementeerd hebt.
Voorbeeld
In onderstaande voorbeeldsessie gaan we ervan uit dat het bestand eiwitten.txt zich in de huidige directory bevindt.
>>> unipept = eiwitDB() >>> unipept.peptideToevoegen('PROT0001', 'ECESAWK') >>> unipept.peptiden {'ECESAWK': {'PROT0001'}} >>> unipept.peptideToevoegen('PROT0002', 'WHK') Traceback (most recent call last): AssertionError: ongeldige peptide >>> unipept.peptideToevoegen('PROT0002', 'ESHLSTLAVQENEIG') Traceback (most recent call last): AssertionError: ongeldige peptide >>> unipept.peptideToevoegen('PROT0002', 'NWAQNAKIGGADWDCVCR') Traceback (most recent call last): AssertionError: ongeldige peptide >>> unipept.eiwitToevoegen('PROT0002', 'HAEWTDNQCCPVLKECESAWKYEMWQHPGEQHKRRRYEMWQHPGEQHKPCHSHTKVWKRY') >>> unipept.peptiden {'ECESAWK': {'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'YEMWQHPGEQHK': {'PROT0002'}} >>> unipept.eiwitToevoegen('PROT0003', 'NRRPCHSHTKECESAWKNRPCHSHTKKPCHSHTKKNRKVWKIPPFFW') >>> unipept.peptiden {'ECESAWK': {'PROT0003', 'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'YEMWQHPGEQHK': {'PROT0002'}} >>> unipept.eiwitToevoegen('PROT0004', 'YEMWQHPGEQHKECESAWKVPYCGFITRPCHSHTKECESAWK') >>> unipept.peptiden {'ECESAWK': {'PROT0004', 'PROT0003', 'PROT0002', 'PROT0001'}, 'PCHSHTK': {'PROT0004', 'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'VPYCGFITR': {'PROT0004'}, 'YEMWQHPGEQHK': {'PROT0004', 'PROT0002'}} >>> unipept.identificeer(['VPYCGFITR']) ['PROT0004'] >>> unipept.identificeer({'ECESAWK', 'PCHSHTK'}) ['PROT0002', 'PROT0003', 'PROT0004'] >>> unipept.identificeer(('YEMWQHPGEQHK', 'ECESAWK', 'PCHSHTK')) ['PROT0002', 'PROT0004'] >>> unipept.identificeer({'PCHSHTK', 'VPYCGFITR'}) ['PROT0004'] >>> unipept.eiwittenToevoegen('eiwitten.txt') >>> unipept.peptiden {'ECESAWK': {'PROT0005', 'PROT0004', 'PROT0003', 'PROT0002', 'PROT0001'}, 'VCEFPWFPMLINDVCR': {'PROT0007'}, 'VPYCGFITR': {'PROT0005', 'PROT0004'}, 'YEMWQHPGEQHK': {'PROT0006', 'PROT0005', 'PROT0004', 'PROT0002'}, 'PCHSHTK': {'PROT0006', 'PROT0005', 'PROT0004', 'PROT0003', 'PROT0002'}, 'HAEWTDNQCCPVLK': {'PROT0002'}, 'CSFHCLEK': {'PROT0006'}, 'AFNYMMPNTK': {'PROT0006'}, 'AYDDEVASFPGCMMATK': {'PROT0007', 'PROT0006'}, 'FIPYYPIYSR': {'PROT0006'}, 'TLCHETMR': {'PROT0005'}, 'HTPNYGVMWMFMNEWMSYDR': {'PROT0006', 'PROT0005'}, 'CDQMHVFDIYMIAIACSWGGPPSLTK': {'PROT0007'}, 'FGHSMTR': {'PROT0005'}} >>> unipept.identificeer(('YEMWQHPGEQHK', 'VPYCGFITR', 'ECESAWK')) ['PROT0004', 'PROT0005'] >>> unipept.identificeer(('PCHSHTK', 'AYDDEVASFPGCMMATK')) ['PROT0006'] >>> unipept.identificeer(['NEGNLNVMK']) []
Bronnen
| Added by: | Peter Dawyndt |
| Date: | 2013-01-02 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |


 RSS
RSS