| Submit | All submissions | Best solutions | Back to list |
PROG0523 - DNA vector |
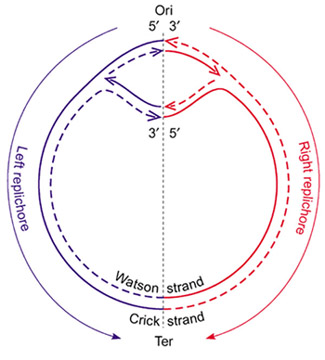
The genome of most bacteria consists of a single circular molecule of DNA. DNA replication — the process of producing two identical replicas from one original DNA molecule — is initiated at a particular sequence in the genome called the origin of replication, and proceeds from this point simultaneous in both directions (see figure below). The replication process ends at a position in the genome called the terminus of replication.
The specific structure of the origin of replication varies somewhat from species to species, but all share some common characteristics such as high AT content (adenine and thymine are easier to separate because they form only two hydrogen bonds whereas guanine and cytosine form three). The origin of replication binds the pre-replication complex, a protein complex that recognizes, unwinds, and begins to copy DNA.
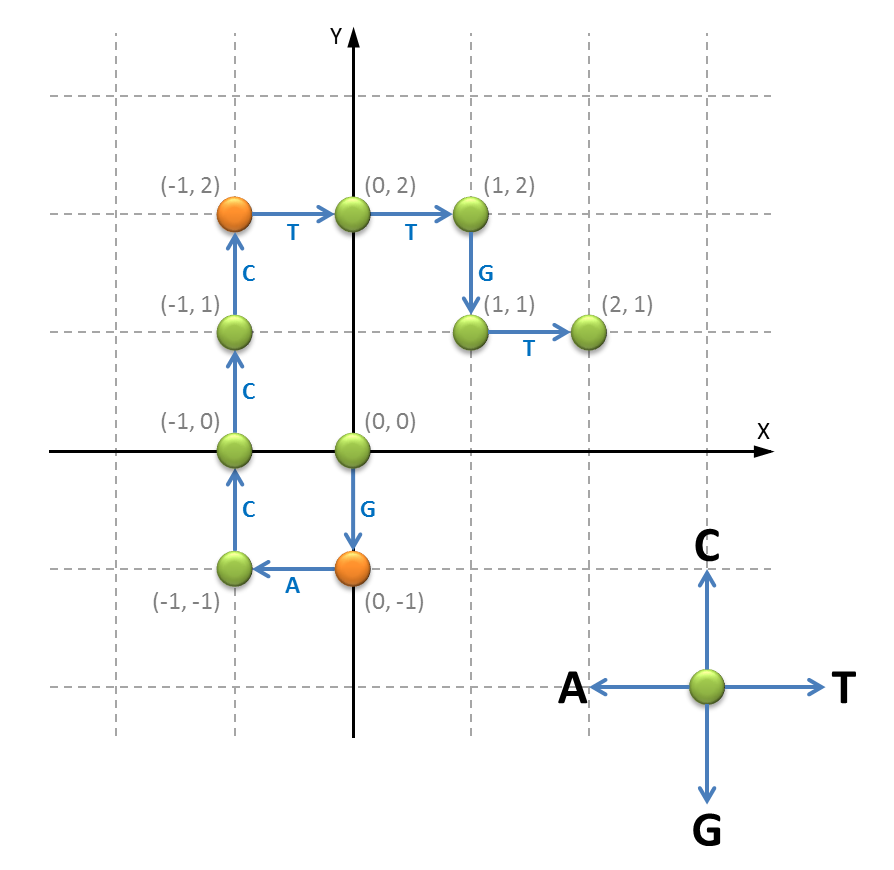
A simple procedure to determine the position of the origin and terminus of replication in a genome sequence, makes us of a vectorial representation of the DNA molecule. This vector is constructed as a list of $(x, y)$-coordinates ($x, y \in \mathbb{N}$) that starts at the point $(0, 0)$ in the origin. For each successive base in the DNA sequence a neighbouring point is visited: the left neighbour for base A, the right neighbour for base T, the upstairs neighbour for base C and the downstairs neighbour for base G. The neighbouring points are always at distance one from the previous point. The figure below shows a graphical display of the vectorial representation of the DNA sequence GACCCTTGT.
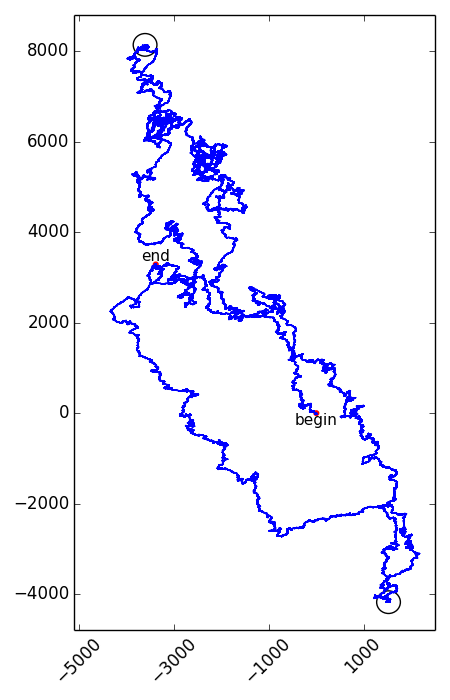
The positions in the vectorial representation where the $y$-coordinate for the first time reaches its maximal and minimal value, correspond to the positions of the origin and terminus of replication on the genome. However, which of the two points is the origin and which one is the terminus can not be determined unambiguously. Below you see an example of the vectorial representation of the complete genome sequence of Haemophilus influenzae strain Rd (L42023), where the positions of the origin and terminus are indicated using black circles.
Assignment
In this assignment, DNA sequences are represented as strings string that only consists of the uppercase letters A, C, G and T. Your are asked to:
- Write a function vector that takes a DNA sequence. The function must return the vectorial representation of the given DNA sequence.
- Use the function vector to write a function replication that takes a DNA sequence as its argument. The function must return a tuple of two integers, that indicate the positions in the vectorial representation of the DNA sequence where the $y$-coordinate for the first time reaches its maximal — resp. minimal — value. The positions in the vectorial representation are increasingly indexed starting from zero.
- Write a function sequence that takes the vectorial representation of a DNA sequence. The function must return the DNA sequence that corresponds to the given vectorial representation.
Example
>>> vector('GACCCTTGT') [(0, 0), (0, -1), (-1, -1), (-1, 0), (-1, 1), (-1, 2), (0, 2), (1, 2), (1, 1), (2, 1)] >>> vector('CTGGGGTAA') [(0, 0), (0, 1), (1, 1), (1, 0), (1, -1), (1, -2), (1, -3), (2, -3), (1, -3), (0, -3)] >>> replicatie('GACCCTTGT') (5, 1) >>> replicatie('CTGGGGTAA') (1, 6) >>> sequentie([(0, 0), (0, -1), (-1, -1), (-1, 0), (-1, 1), (-1, 2), (0, 2), (1, 2), (1, 1), (2, 1)]) 'GACCCTTGT' >>> sequentie([(0, 0), (0, 1), (1, 1), (1, 0), (1, -1), (1, -2), (1, -3), (2, -3), (1, -3), (0, -3)]) 'CTGGGGTAA'
References
- Lobry JR (1996). A simple vectorial representation
of DNA sequences for the detection of replication origins in bacteria. Biochimie
78, 323-326.

- Mackiewicz P, Mackiewicz D,
Kowalczuk M, Cebrat S (2001). Flip-flop around the
origin and terminus of replication in prokaryotic genomes. Genome
Biology 2(12).

Het genoom van de meeste bacteriën bestaat uit één enkele circulaire DNA-molecule. DNA-replicatie — het proces waarbij DNA verdubbeld wordt — start op een bepaalde plaats in het genoom die in het Engels de origin of replication (kortweg ori) genoemd wordt, en wordt vanaf die plaats in twee richtingen tegelijkertijd uitgevoerd (zie onderstaande figuur). Het verdubbelingsproces eindigt op de plaats in het genoom die in het Engels de terminus of replication genoemd wordt.
De specifieke structuur van de origin en terminus of replication verschilt van soort tot soort, maar er zijn enkele gemeenschappelijke eigenschappen zoals een hoge graad van AT (adenine en thymine zijn makkelijker van elkaar te scheiden omdat ze slechts twee waterstofbindingen vormen, terwijl guanine en cytosine er drie vormen). De origin of replication bindt ook met het prereplicatiecomplex, een eiwitcomplex dat DNA herkent, afwikkelt en begint te kopiëren.
Een eenvoudige manier om de positie van de origin en terminus of replication in een genoomsequentie te vinden, maakt gebruik van een vectorvoorstelling van de DNA-molecule. Deze vector wordt opgebouwd als een lijst van $(x, y)$-coördinaten ($x, y \in \mathbb{N}$) die start bij het punt $(0, 0)$ in de oorsprong. Voor elke opeenvolgende base in de DNA-sequentie wordt dan naar een buurpunt gesprongen: voor base A naar de linkerbuur, voor base T naar de rechterbuur, voor base C naar de bovenbuur en voor base G naar de onderbuur. De buren liggen telkens op afstand één van het vorige punt. Hieronder zie je bijvoorbeeld de vectorvoorstelling van de DNA-sequentie GACCCTTGT grafisch weergegeven.
De posities in de vectorvoorstelling waar de $y$-coördinaat voor het eerst zijn maximale en minimale waarde bereikt, corresponderen met de posities van de origin en terminus op het genoom. Er kan echter niet eenduidig vastgelegd worden welke van de twee posities de origin en welke de terminus is. Hieronder zie je bijvoorbeeld de vectorvoorstelling van de volledige genoomsequentie van Haemophilus influenzae stam Rd (L42023), waarbij de posities van de origin en terminus of replication worden aangegeven met zwarte cirkels.
Opgave
In deze opgave stellen we DNA-sequenties voor als strings die enkel bestaan uit de hoofdletters A, C, G en T. Gevraagd wordt:
- Schrijf een functie vector waaraan een DNA-sequentie moet doorgegeven worden. De functie moet de vectorvoorstelling van de gegeven DNA-sequentie teruggeven.
- Gebruik de functie vector om een functie replicatie te schrijven waaraan een DNA-sequentie moet doorgegeven worden. De functie moet een tuple van twee gehele getallen teruggeven, die de posities in de vectorvoorstelling van de DNA-sequentie aangeven waar de $y$-coordinaat voor het eerst zijn maximale — resp. minimale — waarde bereikt. De posities in de vectorvoorstelling worden oplopend genummerd vanaf nul.
- Schrijf een functie sequentie waaraan de vectorvoorstelling van een DNA-sequentie moet doorgegeven worden. De functie moet de DNA-sequentie die correspondeert met de gegeven vectorvoorstelling teruggeven.
Voorbeeld
>>> vector('GACCCTTGT') [(0, 0), (0, -1), (-1, -1), (-1, 0), (-1, 1), (-1, 2), (0, 2), (1, 2), (1, 1), (2, 1)] >>> vector('CTGGGGTAA') [(0, 0), (0, 1), (1, 1), (1, 0), (1, -1), (1, -2), (1, -3), (2, -3), (1, -3), (0, -3)] >>> replicatie('GACCCTTGT') (5, 1) >>> replicatie('CTGGGGTAA') (1, 6) >>> sequentie([(0, 0), (0, -1), (-1, -1), (-1, 0), (-1, 1), (-1, 2), (0, 2), (1, 2), (1, 1), (2, 1)]) 'GACCCTTGT' >>> sequentie([(0, 0), (0, 1), (1, 1), (1, 0), (1, -1), (1, -2), (1, -3), (2, -3), (1, -3), (0, -3)]) 'CTGGGGTAA'
Bronnen
- Lobry JR (1996). A simple vectorial representation
of DNA sequences for the detection of replication origins in bacteria. Biochimie
78, 323-326.

- Mackiewicz P, Mackiewicz D,
Kowalczuk M, Cebrat S (2001). Flip-flop around the
origin and terminus of replication in prokaryotic genomes. Genome
Biology 2(12).

| Added by: | Peter Dawyndt |
| Date: | 2014-12-28 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS